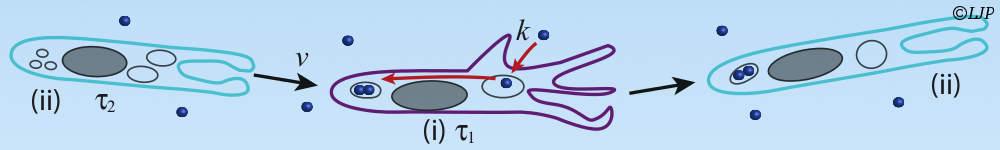
Morphogenèse
André Estevez-Torres, Jean-Christophe Galas, Baptiste Blanc, Nicolas Lobato-Dauzier, Romain Leroux
Former members: Guillaume Sarfati, Marc Van Der Hofstadt, Yuliia Vyborna, Anis Senoussi, Shunichi Kashida, Georg Urtel, Anton Zadorin, Jonathan Lee Tin Wah, Adrian Zambrano
News: Nous venons d'obtenir un finacement ANR (LiliMat - Life-Like Materials, 2022-2026) ainsi qu'un financement PEPR (MoleculArxiv - DNA synthesis for data storage). Nous avions obtenu en 2018 une ERC consolidator grant intitulée 'Matière molle metabolique inspirée du vivant'.
Vous trouverez des informations plus détaillées sur notre page web dédiée: http://ljpmorpho.free.fr
Nous assemblons des systèmes biochimiques à base d’acides nucléiques synthétiques (ADN et ARN) qui s'auto-organisent dans l'espace dans des formes bien définies. Nous nous intéressons notamment aux processus d'auto-organisation hors équlibre, c'est à dire qui ont besoin de consommer de l'énergie chimique pour rester ordonnés, à l'instar des processus de réaction-diffusion. Nous menons cette approche biomimétique avec deux objectifs. D’un côté, en étudiant des systèmes moléculaires simples qui émulent leurs analogues biologiques nous espérons pouvoir mieux comprendre l’émergence de l'ordre dans le vivant, particulièrement lors de la morphogénèse. D’un autre côté, ces systèmes dynamiques moléculaires peuvent être vus comme un nouveau type de matériau avec des propriétés qui sont classiques dans les organismes vivants mais rares dans les matériaux synthétiques: des matériaux capables de se construire par eux mêmes, de bouger et de "parler" avec le vivant.
Pour ce faire nous utilisons essentiellement des systèmes reposant sur des réactions d'hybridation d'acides nucléiques parce que leur réactivité peut être prédite facilement en utilisant les règles d'appariement dites Watson et Crick. Plus précisément, nous travaillons avec les systèmes expérimentaux suivants (principalement les 1 et 2) qui sont puissants et complémentaires :
- 1) La boîte à outils PEN DNA, un langage de programmation moléculaire fondé sur des courts brins d'ADN et trois enzymes qui peut être tenu hors équilibre pendant des dizaines d'heures.
- 2) Le système microtubule/kinesine, une matière active polyvalente capable de générer des transductions chimio-mécaniques
- 3) Des systèmes des transcription-translation qui fonctionnent en dehors d'une cellule vivante et qui nous premettent d'étudier la dynamique de réseaux de régulations génétique in vitro.
- 4) Des nanostructures d'ADN auto-assemblées selon la méthode de l'origami d'ADN.
En plus de ces technologies moléculaires nous développons des techniques microfluidiques et de microstructuration pour le contrôle spatial et temporel des systèmes réactifs décrits ci-dessus.
Publications récentes
- Sarfati G, Maitra A, Voituriez R, Galas J-C, Estévez-Torres A, Crosslinking and depletion determine spatial instabilities in cytoskeletal active matter, Soft Matter, 2022
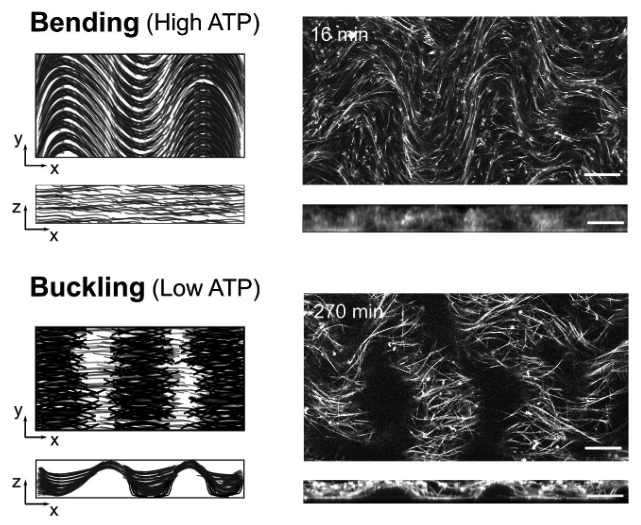
- Galas J-C, Estévez-Torres A, Van Der Hofstadt M, Long-Lasting and Responsive DNA/Enzyme-Based Programs in Serum-Supplemented Extracellular Media, ACS Synthetic Biology, 2022
- del Junco C, Estévez-Torres A, Maitra A, Front speed and pattern selection of a propagating chemical front in an active fluid, Physical Review E, 2022
- Senoussi A, Galas JC, Estévez-Torres A, Programmed mechano-chemical coupling in reaction-diffusion active matter, Science Advances, 2021

- Vyborna Y, Galas JC, Estévez-Torres A, DNA-Controlled Spatiotemporal Patterning of a Cytoskeletal Active Gel, JACS, 2021
- Senoussi A, Vyborna Y, Berthoumieux H, Galas JC, Estévez-Torres A, Learning from Embryo Development to Engineer Self-organizing Materials chapter in 'Out-of-Equilibrium (Supra)molecular Systems and Materials' book (Editor: Nicolas Giuseppone, Andreas Walther), Wiley, 2021
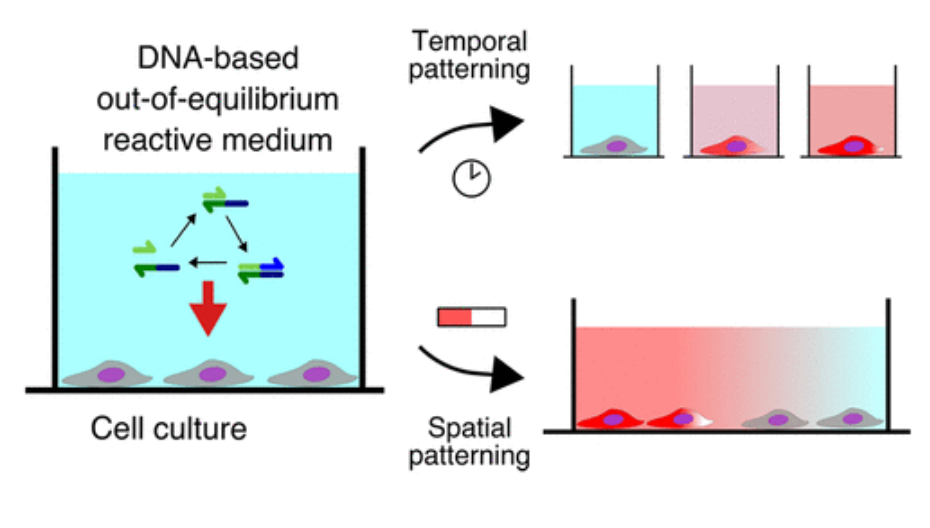
- Van Der Hofstadt M, Galas J-C, Estévez-Torres A, Spatiotemporal Patterning of Living Cells with Extracellular DNA Programs, ACS Nano, 2021
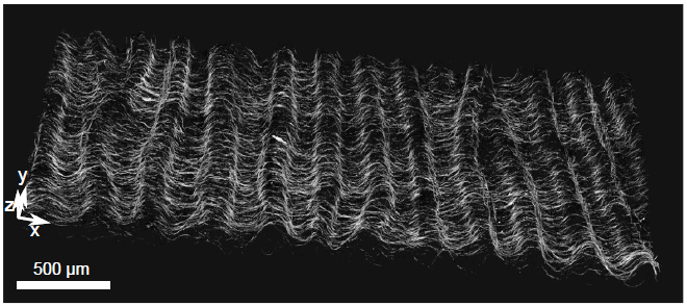
- Senoussi A, Kashida S, Voituriez R, Galas J-C, Maitra A, Estévez-Torres A, Tunable corrugated patterns in an active gel sheet, PNAS, 2019 Preprint on arxiv
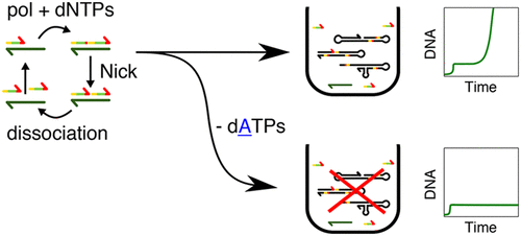
- Urtel G, Van Der Hofstadt M, Galas J-C, Estévez-Torres A,
rEXPAR: an isothermal amplification scheme that is robust to autocatalytic parasites,
ACS Biochemistry, 2019 Preprint on arxiv
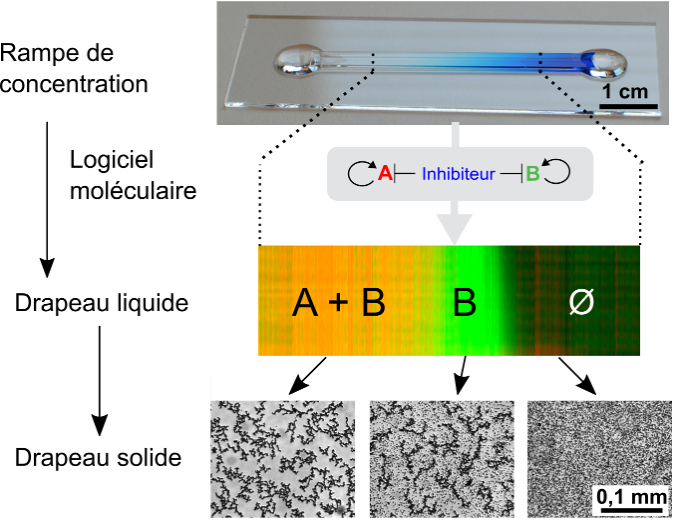
- A. Zadorin, Y. Rondelez, G. Gines, V. Dilhas, G. Urtel, A. Zambrano, J.-C. Galas, A. Estevez-Torres, Synthesis and materialization of a reaction-diffusion French flag pattern, Nature chem., 2017, doi:10.1038/nchem.2770. Preprint on arxiv
- G. Gines, A. Zadorin, J.-C Galas, T. Fujii, A. Estevez-Torres, Y. Rondelez, Microscopic agents programmed by DNA circuits, Nature nano, 2017, doi:10.1038/nnano.2016.299
- Lee Tin Wah J, David C, Rudiuk S, Baigl D, Estévez-Torres, A, Observing and Controlling the Folding Pathway of DNA Origami at the Nanoscale, ACS nano, 2016.
- Estevez-Torres A, Biophysics: The expressionist movement, Nature Physics, 2015. News and views.
- Zambrano A, Zadorin AS, Rondelez Y, Estevez-Torres A, Galas JC, Pursuit-and-evasion reaction- diffusion waves in microreactors with tailored geometry, J. Phys. Chem. B, 119(17) :5349-5355, 2015.
- Zadorin AS, Rondelez Y, Galas J-C, & Estevez-Torres A, Synthesis of programmable reaction-diffusion fronts using DNA catalyzers. Phys. Rev. Lett. 2015 114(6). Highlighted in Nature nanotech, Physics and Chemistry world.
Plus d'informations sur: http://ljpmorpho.free.fr
Et n'hésitez pas à consulter notre canal youtube