MAI 2017
Inspirés par un processus mis en jeu au tout début du développement embryonnaire, des chimistes et des physiciens ont conçu une solution aqueuse qui s'auto-organise en une structure à trois bandes chimiquement distinctes.
Le modèle du « drapeau français » est l'une des représentations conceptuelles les plus connues de la spatialisation en biologie. Il a été introduit en 1969 par le biologiste britannique L. Wolpert dans le but d'expliquer comment, à partir d'un gradient maternel initialement présent, un embryon pouvait se segmenter en trois bandes chimiquement distinctes qui vont devenir la tête, le corps et les jambes. Ce processus de spatialisation, qui constitue un tout premier pas vers l'acquisition d'une forme définitive, repose notamment sur un « logiciel moléculaire » composé de réseaux de réactions chimiques qui confèrent aux cellules leurs étonnantes capacités à traiter de l'information, en particulier celle de pouvoir mesurer un niveau de concentration. Reproduire ce mécanisme naturel de manière synthétique est un enjeu majeur tant du point de vue de notre compréhension des processus d'auto-organisation dans le vivant que pour concevoir de nouveaux matériaux. Une équipe de chercheurs du Laboratoire Jean Perrin (CNRS / Univ. Pierre et Marie Curie), du Laboratoire Gulliver (CNRS / ESPCI), du LIMMS (CNRS / U. Tokyo) et de l'Université de Munich a réussi, pour la première fois, à programmer une solution aqueuse pour qu'elle s'auto-organise en « drapeau français ». Ce travail est publié dans la revue Nature Chemistry.
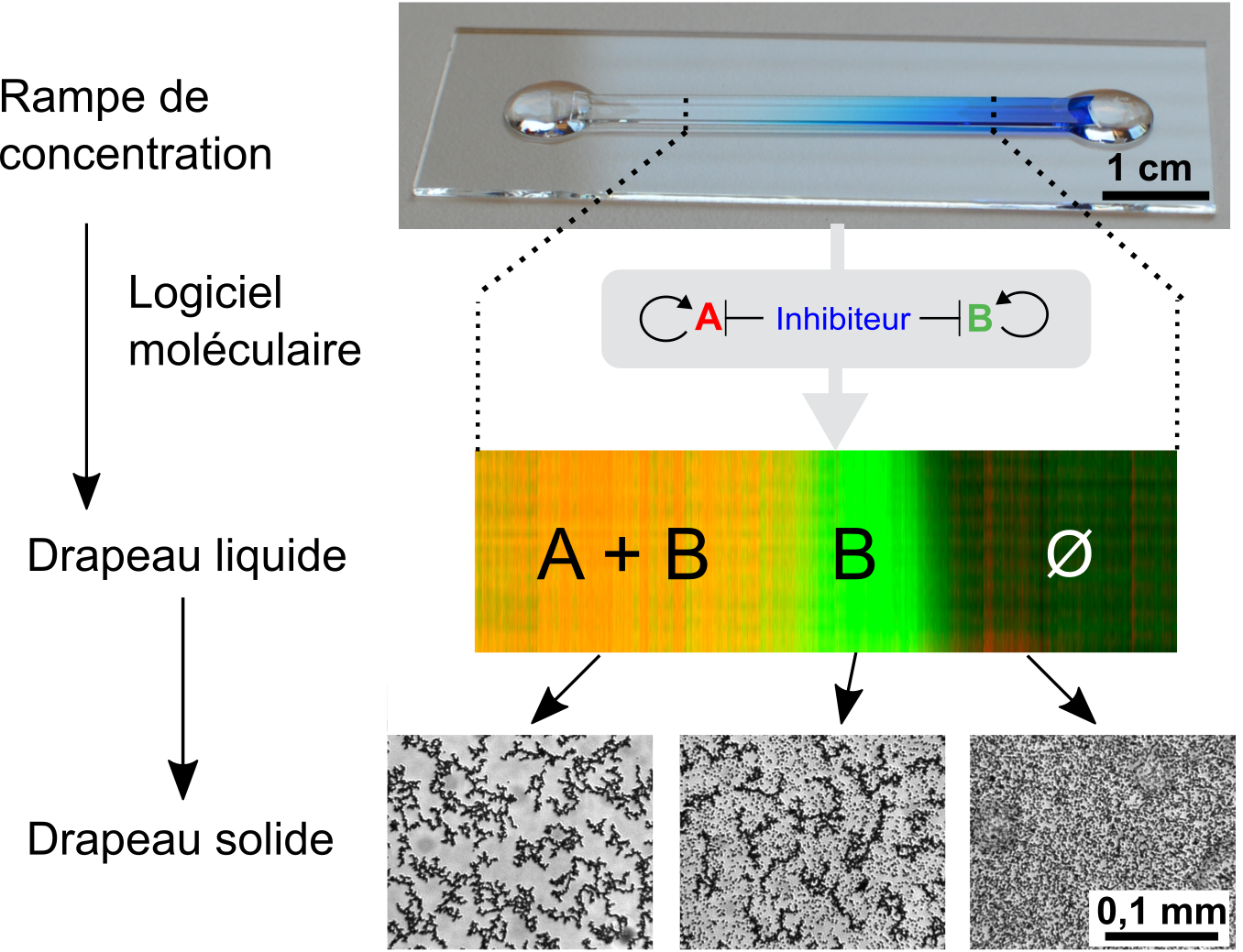
Une rampe de concentration dans un micro-canal interprétée par un logiciel moléculaire fait apparaître une carte chimique à trois bandes. D'abord liquide, cette carte se solidifie à l'aide de microbilles. (C) LJP J.-C. Galas et A. Estevez-Torres.
Pour obtenir ces résultats, les chercheurs ont créé un « logiciel moléculaire » constitué de réactions chimiques. Si l'évolution a doté les cellules d'un « logiciel » puissant, en concevoir une version synthétique est un exploit en soi, tant il est difficile de contrôler la réactivité moléculaire. Pour contourner ce problème, ils ont utilisé de petites molécules d'ADN réagissant avec trois enzymes. Le « logiciel » est composé d'un ADN activateur et d'un ADN inhibiteur et n'a que deux états possibles: en deçà d'une concentration seuil d'inhibiteur, l'activateur est produit, au dessus il est détruit. En générant une rampe de concentration d'inhibiteur dans un tuyau de la taille d'une allumette –remplie d'eau, d'ADN et d'enzymes– l'activateur est produit de façon continue en bas de la rampe, qu'il remonte par diffusion en s'arrêtant lorsque la concentration d'inhibiteur devient trop importante. Ceci forme une carte chimique à deux bandes. La structure en « drapeau français » est obtenue en associant deux couples activateur/inhibiteur présentant des seuils différents, qui génèrent alors trois régions chimiquement distinctes. Ce drapeau liquide est alors solidifié à l'aide de petites billes micrométriques qui peuvent se coller entre elles en fonction de la composition des bandes chimiques.
Au delà de fournir un outil précieux pour la compréhension du fonctionnement des programmes moléculaires du vivant, ce travail ouvre la voie vers la conception de matériaux « morphogénétiques », c'est à dire capables de s'auto-construire en suivant les instructions d'un programme moléculaire, à l'image du développement d'un embryon.
En savoir plus
Synthesis and materialization of a reaction-diffusion French flag pattern, A. Zadorin, Y. Rondelez, G, Gines, V. Dilhas, G. Urtel, A. Zambrano, J.-C. Galas, and A. Estevez-Torres, Nat. Chem.
Contact chercheur
J.-C. Galas (jean-christophe.galas@upmc.fr)
A. Estevez-Torres (andre.estevez-torres@upmc.fr)
Informations complémentaires
Laboratoire Jean Perrin (http://www.labos.upmc.fr/ljp/?article13)