2024
Biofilm bactérien multi-espèces en Micro-spectroscopie Raman
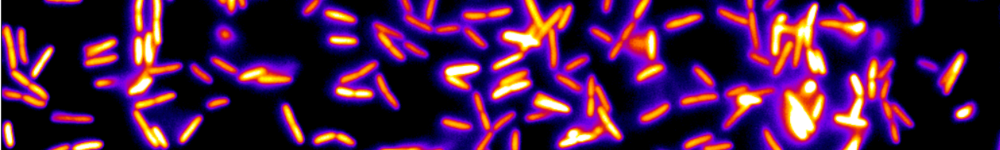
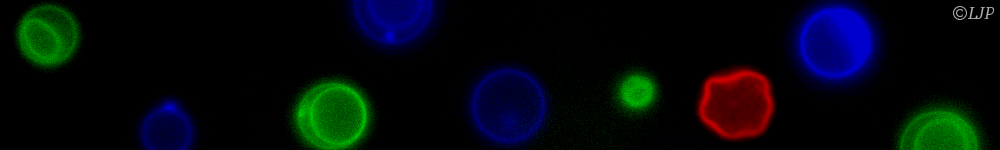
Nous proposons d’étudier l’assemblage de la matrice extracellulaire d’un biofilm bactérien multi-espèces par micro-spectroscopie Raman. Ce projet s’appuie sur des travaux récents menés avec Nelly Henry au LJP où un modèle expérimental robuste de biofilm multi-espèces vient d’être établi et caractérisé et sur les innovations technologiques récentes introduites en Raman compressif au LKB par Hilton Barbosa de Aguiar. Cette collaboration interdisciplinaire permettra de (i) réaliser la première micro-analyse spectroscopique in situ d’une communauté vivante en croissance (ii) de comprendre l’assemblage d’un modèle vivant contrôlé de communauté écologique — le biofilm bactérien multi-espèces. Plus de détails ici
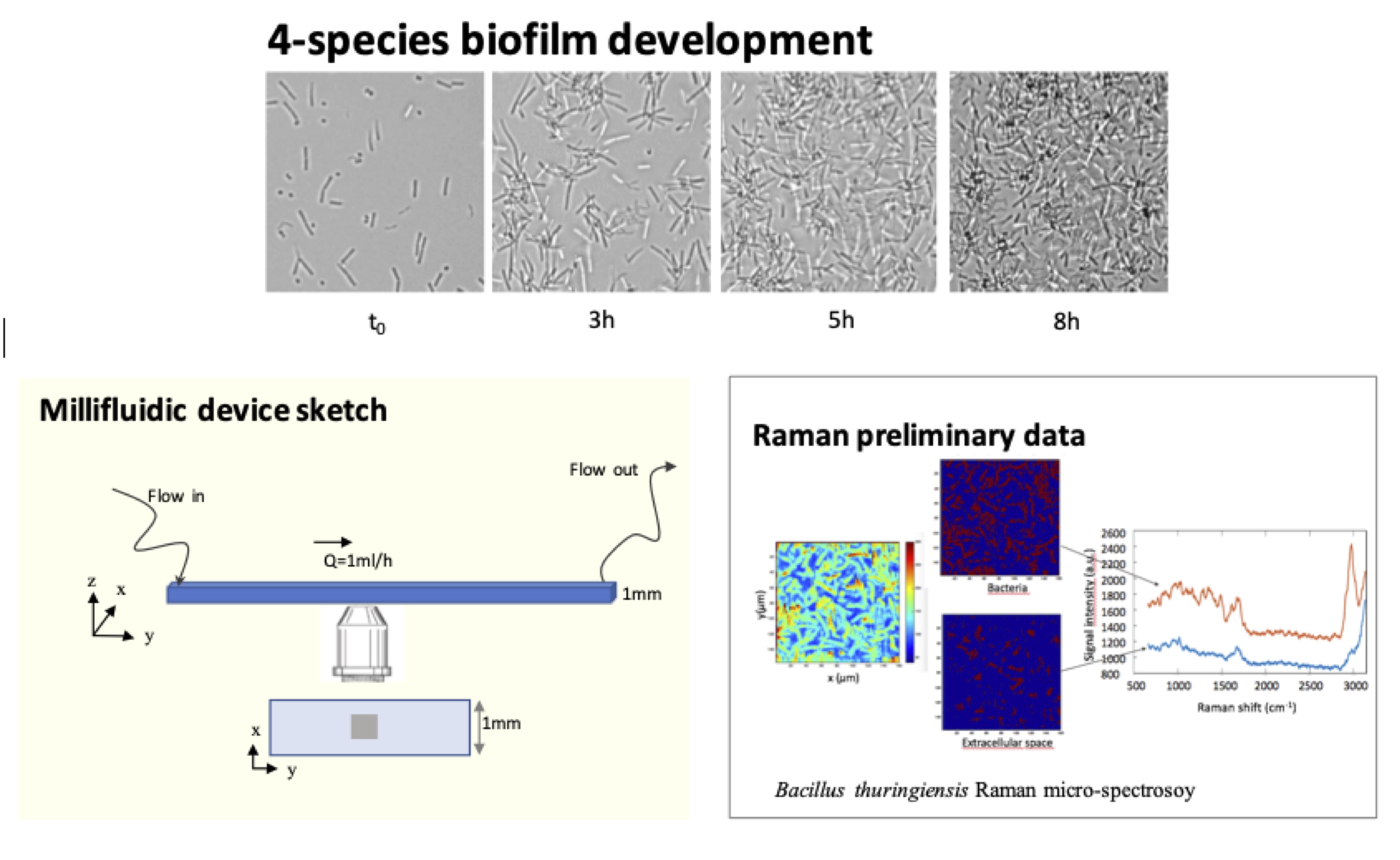
Ce projet s'inscrit dans le nouveau programme interdisciplinaire i-Bio. Il sera co-encadré par Nelly Henry - Laboratoire Jean Perrin (CNRS, SU, Campus Jussieu, Paris 75005) et Hilton B. de Aguiar (LKB, CNRS, ENS, Campus ENS Ulm, Paris F-75005). full iBio project
Profil du candidat: Formation principale en physique ou physico-chimie avec un fort intérêt pour la biologie. Un étudiant de master 2 à l'interface de ces disciplines serait très apprécié. Cependant, une forte motivation pour les systèmes biologiques sera également considérée favorablement pour un candidat sans formation en biologie.
Contact: nelly.henry@sorbonne-universite.fr et h.aguiar@phys.ens.fr
Références:
1. P. Thomen, J. D. P. Valentin, A. F. Bitbol, N. Henry, Spatiotemporal pattern formation in E.coli biofilms explained by a simple physical energy balance. Soft Matter 16, 494-504 (2020)
2. Monmeyran et al., The inducible chemical-genetic fluorescent marker FAST outperforms classical fluorescent proteins in the quantitative reporting of bacterial biofilm dynamics. Sci Rep 8, 10336 (2018)
3. P. Thomen et al., Bacterial biofilm under flow: First a physical struggle to stay, then a matter of breathing. PLoS ONE 12, e0175197 (2017)
4. O. Galy et al., Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophysical journal 5, 1400-1408 (2012).
5. S. H. Donaldson Jr., H. B. de Aguiar. Molecular Imaging of Cholesterol and Lipid Distributions in Model Membranes. J. Phys. Chem. Lett. 9 1528 (2018)
6. B. Sturm, F. Soldevila, E. Tajahuerce, S. Gigan, H. Rigneault, H. B. de Aguiar. High-sensitivity high-speed compressive spectrometer for Raman imaging. ACS Photonics 6, 1409–1415 (2019)
7. F. Soldevila, J. Dong, E. Tajahuerce, S. Gigan, H. B. de Aguiar. Fast compressive Raman bio-imaging via matrix completion. Optica 6, 341–346 (2019)